KerasでMNIST CNNその2
前回は MNIST のデータセットを CNN で認識する 例題を実践した。とりあえず文字認識はできたものの、ネットワークモデルの決め方がよくわからなかったので今回追加で実験してみる。
モデルのバリエーションを試すので、まずはモデルの可視化からやってみる。
1. 前準備
といきたいところだが、まずは環境の用意が必要。 モデルの可視化をする上で Graphviz と pydot の両方をインストールする必要がある。
私は Keras を Docker 上で動かしているので、Docker イメージの方から作り直した。
DockerFile の内容は Keras の環境構築をした その2 へ加筆したのでそちらを参照。
あと、ライブラリのインポートを先に済ませておく。
import matplotlib
matplotlib.use("Agg")
import matplotlib.pyplot as plt
import numpy as np
import keras
from keras import backend as K
from keras.datasets import mnist
from keras.models import Model, Sequential
from keras.layers import Dense, Dropout, Flatten
from keras.layers import Conv2D, MaxPooling2D
from keras.utils import plot_model
img_rows, img_cols = 28, 28
num_classes = 10
batch_size = 128
テストデータも読んでおく。
(x_train, y_train), (x_test, y_test) = mnist.load_data()
x_train = x_train.reshape(x_train.shape[0], img_rows, img_cols, 1)
x_test = x_test.reshape(x_test.shape[0], img_rows, img_cols, 1)
x_train = x_train.astype('float32') / 255
x_test = x_test.astype('float32') / 255
y_train = keras.utils.to_categorical(y_train, 10)
y_test = keras.utils.to_categorical(y_test, 10)
では改めて、
2. モデルの可視化
モデルのバリエーションを試すので、まずはモデルの可視化からやってみる。
これも前回と同じモデルをまずは構築する。
model = Sequential()
model.add(Conv2D(32, kernel_size=(3, 3),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(128, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(num_classes, activation='softmax'))
以下の層が積み重なったモデルである。
- 2層の畳み込み層
- 1層のプーリング層
- 2層の全結合層
これを可視化には次のようにする。
plot_model(model, to_file='model1.png', show_shapes=True)
なんか出た。
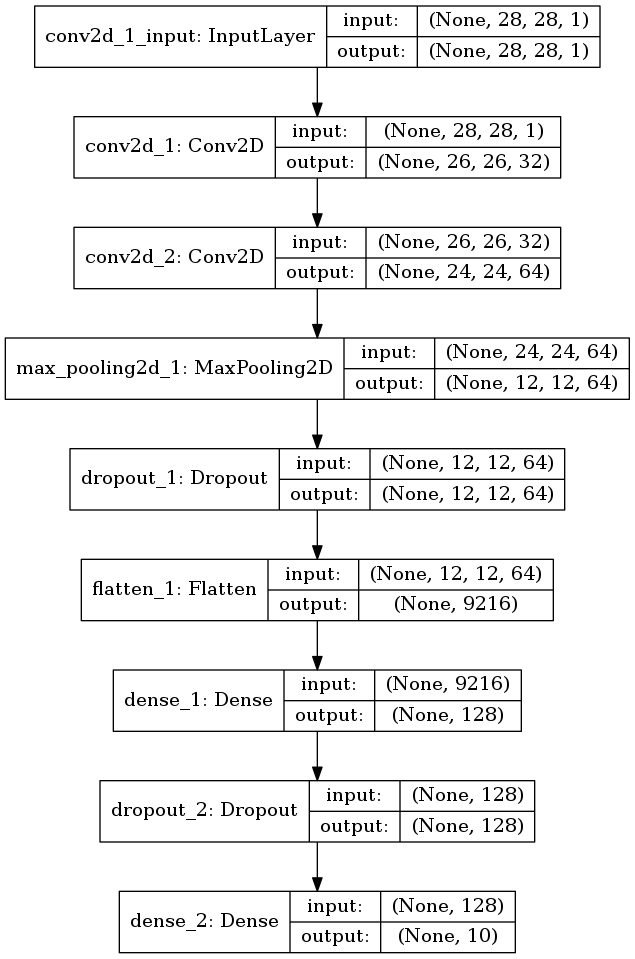
図2.1. CNNのモデル その1
なるほど各層の入出力の次元が見える感じね。 前回手で計算したやつがパッとわかるみたいだ。
今回はこれを使って可視化しながら、モデルのバリエーションを試してみたいと思う。
3. モデルを色々変えてみる
以下をやってみたい。
- 畳み込み層が1層だけだったら
- 畳み込み層が3層だったら
- プーリング層が無かったら
- プーリングのサイズが大きかったら
- 全結合層が1層だけだったら
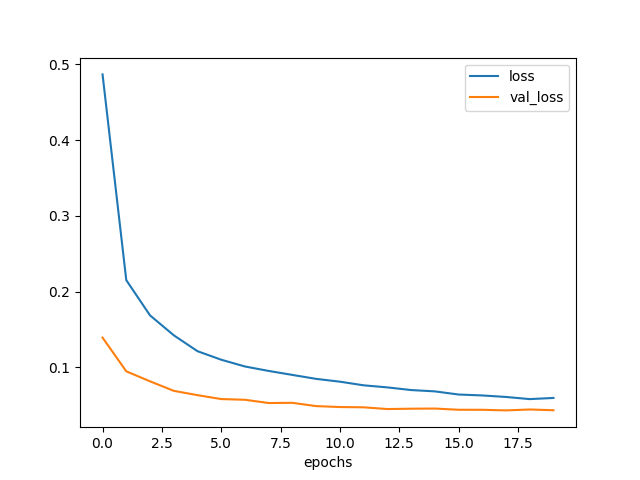
元のモデル
元のモデルの学習曲線を再掲しておく。
もしも畳み込み層が1層だけだったら
2つ目の畳み込み層を落してみる。 代わりに1つ目の畳み込み層のフィルタサイズを大きくして、上層の次元数を合わせる。
モデルはこうなる。
model = Sequential()
model.add(Conv2D(64, kernel_size=(5, 5),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(128, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model2.png', show_shapes=True)
プーリング層以降は元のモデルと完全に一致する。
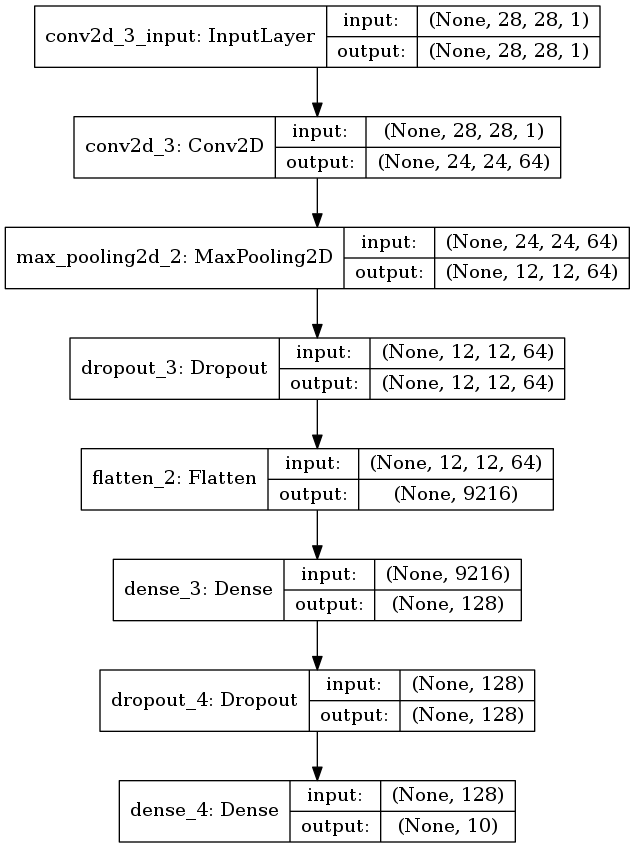
図3.1. 畳み込み層が1層のモデル
訓練させてみる。
model.compile(loss=keras.losses.categorical_crossentropy,
optimizer=keras.optimizers.Adadelta(),
metrics=['accuracy'])
hist = model.fit(x_train, y_train,
batch_size=batch_size,
epochs=20,
validation_split=0.1,
verbose=1)
scores = model.evaluate(x_test, y_test)
print('loss={}, accuracy={}'.format(*scores))
# loss=0.0268098793532, accuracy=0.9914
ん?意外にも好成績か?
学習曲線を描いてみた。
plt.clf()
plt.xlabel('epochs')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['loss'], label='loss')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['val_loss'], label='val_loss')
plt.legend()
plt.savefig('model2-epochs-loss.png')
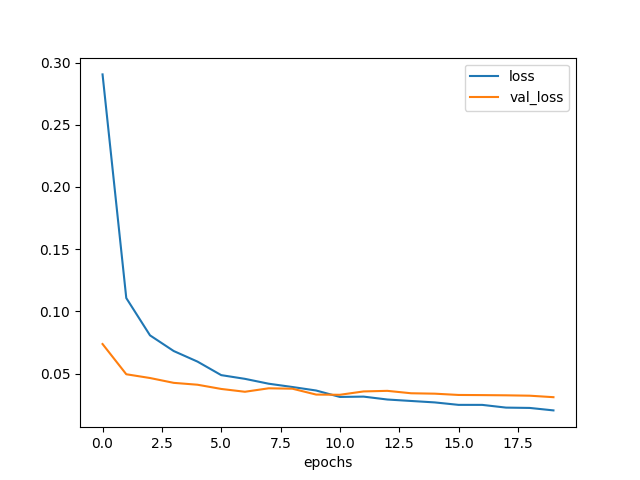
図3.2. 畳み込み層が1層のときの学習曲線
確かに loss は元のモデルよりも早く下がっている。
しかし val_loss は3エポックくらいで下げ止まってしまっていて、
最終的な値は元のモデルと同じ位である。
元のモデルよりも過学習になり易いのかもしれない。
畳み込み層が3層だったら
今度は畳み込み層を増やしてみる。 次元数が変わってしまうが、1つ目の全結合層の重み行列の次元数を減らして対応する。
model = Sequential()
model.add(Conv2D(32, kernel_size=(3, 3),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(128, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model3.png', show_shapes=True)
可視化する。
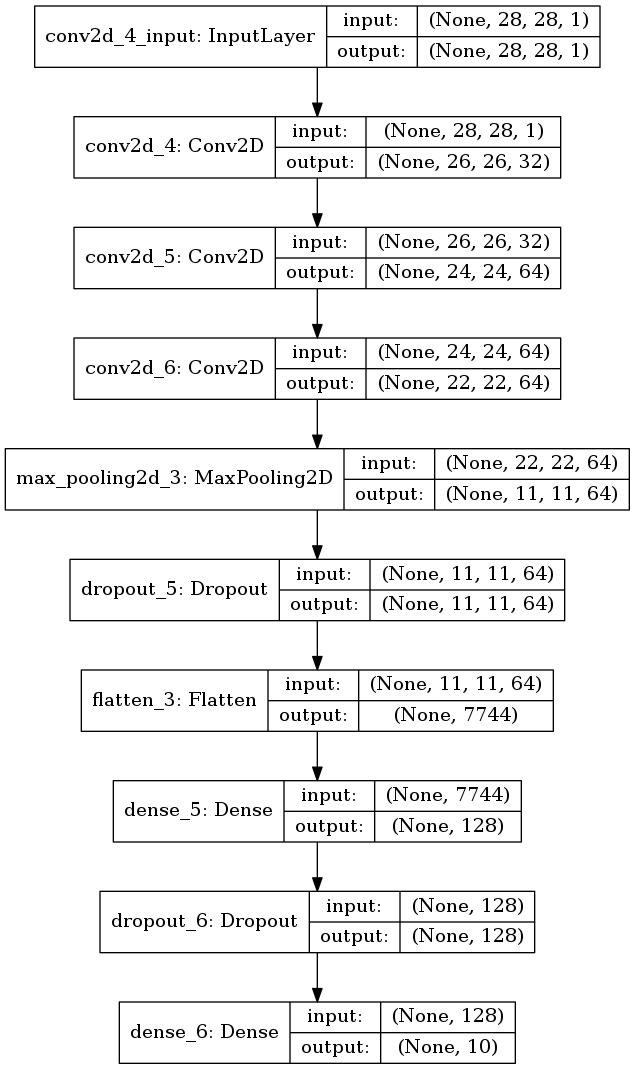
図3.3. 畳み込み層が2層のモデル
訓練と評価する。
model.compile(loss=keras.losses.categorical_crossentropy,
optimizer=keras.optimizers.Adadelta(),
metrics=['accuracy'])
hist = model.fit(x_train, y_train,
batch_size=batch_size,
epochs=20,
validation_split=0.1,
verbose=1)
scores = model.evaluate(x_test, y_test)
print('loss={}, accuracy={}'.format(*scores))
# loss=0.0247306166548, accuracy=0.9926
plt.clf()
plt.xlabel('epochs')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['loss'], label='loss')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['val_loss'], label='val_loss')
plt.legend()
plt.savefig('model3-epochs-loss.png')
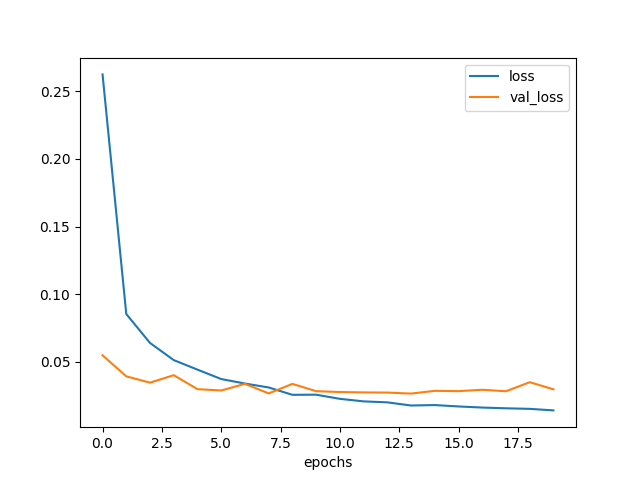
図3.4. 畳み込み層が2層のときの学習曲線
loss はさらに下がったが、 val_loss は途中から上がってしまっている。
まあ前回、2層でこれ以上無いくらい細かい特徴が抽出されてしまっている感があったので フィルタを増やしても効果が無いのは仕方無いかと思う。
プーリング層が無かったら
プーリング層は位置の感度を下げる機能があるらしいので、 MNISTだと有っても無くてもそんなに変らないんじゃないかと思う。
単純にプーリング層だけを抜いたモデルを作った。
model = Sequential()
model.add(Conv2D(32, kernel_size=(3, 3),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(128, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model4.png', show_shapes=True)
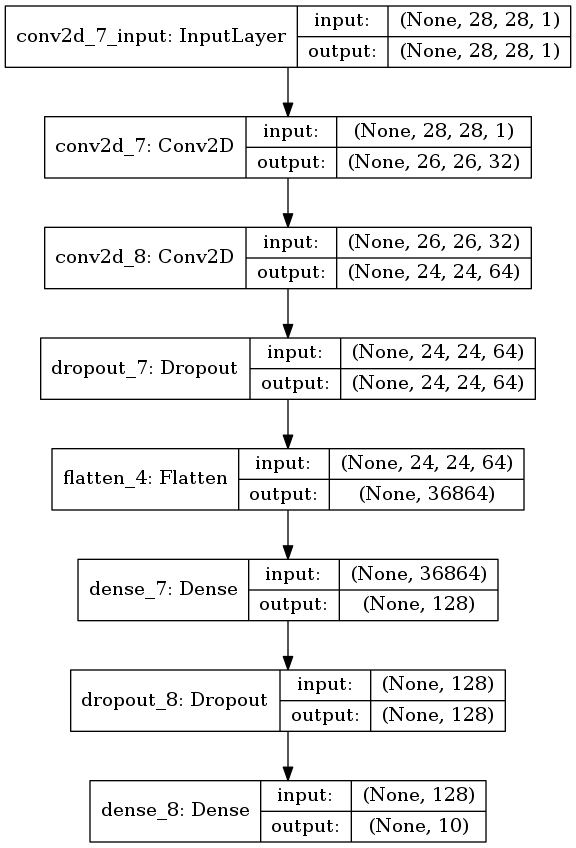
図3.5. プーリング層が無いモデル
訓練と評価。
model.compile(loss=keras.losses.categorical_crossentropy,
optimizer=keras.optimizers.Adadelta(),
metrics=['accuracy'])
hist = model.fit(x_train, y_train,
batch_size=batch_size,
epochs=20,
validation_split=0.1,
verbose=1)
scores = model.evaluate(x_test, y_test)
print('loss={}, accuracy={}'.format(*scores))
# loss=0.0369513304536, accuracy=0.9896
plt.clf()
plt.xlabel('epochs')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['loss'], label='loss')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['val_loss'], label='val_loss')
plt.legend()
plt.savefig('model4-epochs-loss.png')
3エポックくらいから過学習し始めた。
しかも val_loss は底でも 0.04 くらいで、これまでと比べると性能はよくない。
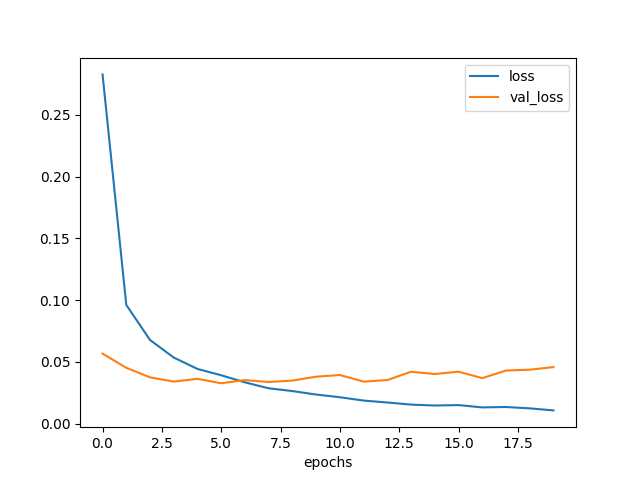
図3.6. プーリング層が無いときの学習曲線
プーリング層の効果はけっこう大きいようだ。
じゃあ逆にプール領域のサイズが大きかったら性能が上るのかもしれない。
プーリング層のプール領域が大きかったら
プール領域のサイズを2倍にしてみた。
model = Sequential()
model.add(Conv2D(32, kernel_size=(3, 3),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(MaxPooling2D(pool_size=(4, 4)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(128, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model5.png', show_shapes=True)
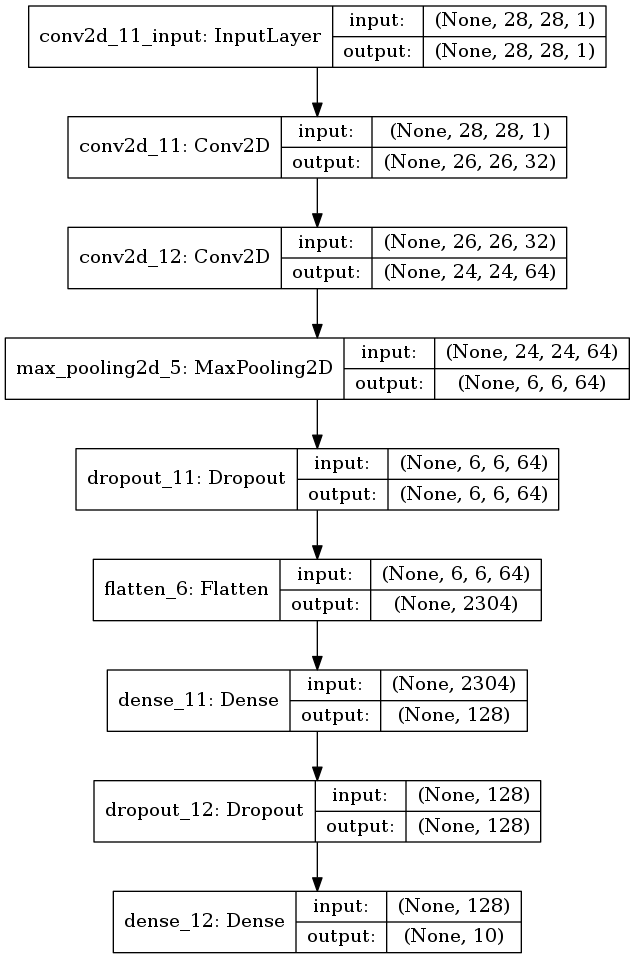
図3.7. プール領域が大きいモデル
評価と訓練。
model.compile(loss=keras.losses.categorical_crossentropy,
optimizer=keras.optimizers.Adadelta(),
metrics=['accuracy'])
hist = model.fit(x_train, y_train,
batch_size=batch_size,
epochs=20,
validation_split=0.1,
verbose=1)
scores = model.evaluate(x_test, y_test)
print('loss={}, accuracy={}'.format(*scores))
# loss=0.0224432536123, accuracy=0.9927
plt.clf()
plt.xlabel('epochs')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['loss'], label='loss')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['val_loss'], label='val_loss')
plt.legend()
plt.savefig('model5-epochs-loss.png')
飽きてきた。
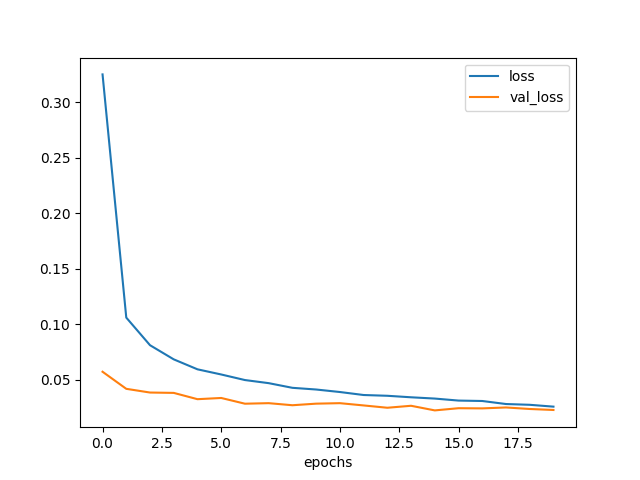
図3.8. プール領域が大きいときの学習曲線
いちおうこのモデルが今回の検証では一番の性能になった。 数字1桁については、データが単純なのでプーリング層は大きめで問題無かったと想像する。 ただしデータに対してプーリング層が大きすぎる場合には、重要な特徴がぼやけてしまい 性能に影響することが予想される。絵とかでは注意した方がいいかもしれない。
たぶん平均プーリングもやっておくのが勤勉というものだと思うのだが、 それほど変わらないと思うのでやらない。
次で最後にする。
全結合層が1層だったら
# Model6
model = Sequential()
model.add(Conv2D(32, kernel_size=(3, 3),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model6.png', show_shapes=True)
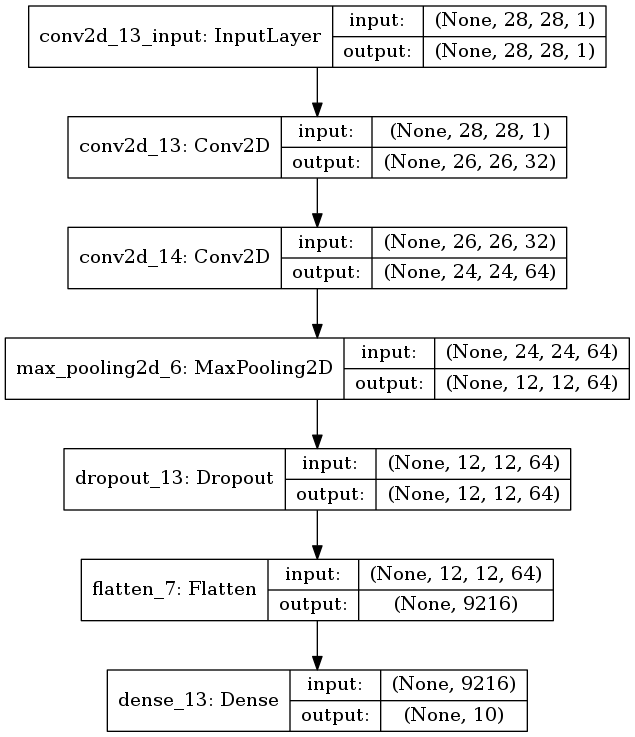
*図3.9. 全結合層が1層だけのモデル
model.compile(loss=keras.losses.categorical_crossentropy,
optimizer=keras.optimizers.Adadelta(),
metrics=['accuracy'])
hist = model.fit(x_train, y_train,
batch_size=batch_size,
epochs=20,
validation_split=0.1,
verbose=1)
scores = model.evaluate(x_test, y_test)
print('loss={}, accuracy={}'.format(*scores))
# loss=0.0355213801761, accuracy=0.9897
plt.clf()
plt.xlabel('epochs')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['loss'], label='loss')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['val_loss'], label='val_loss')
plt.legend()
plt.savefig('model6-epochs-loss.png')
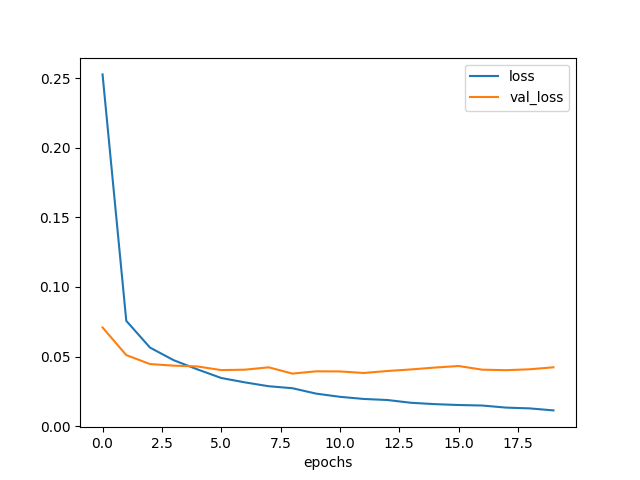
図3.10. 全結合層が1層だけのときの学習曲線
1層だけだと学習しきれなかったみたいだ。
4. まとめ
- Keras では層の増減、次元数の変更あたりがすごく簡単。
- 少しくらいモデルを変えてもテストデータに対する性能が大きく変わらない。
- モデルの組み方により過学習のなり易さが違う。
5. ソースコードぜんぶん
貼っておく。
import matplotlib
matplotlib.use("Agg")
import matplotlib.pyplot as plt
import numpy as np
import keras
from keras import backend as K
from keras.datasets import mnist
from keras.models import Model, Sequential
from keras.layers import Dense, Dropout, Flatten
from keras.layers import Conv2D, MaxPooling2D
from keras.utils import plot_model
img_rows, img_cols = 28, 28
num_classes = 10
batch_size = 128
(x_train, y_train), (x_test, y_test) = mnist.load_data()
x_train = x_train.reshape(x_train.shape[0], img_rows, img_cols, 1)
x_test = x_test.reshape(x_test.shape[0], img_rows, img_cols, 1)
x_train = x_train.astype('float32') / 255
x_test = x_test.astype('float32') / 255
y_train = keras.utils.to_categorical(y_train, 10)
y_test = keras.utils.to_categorical(y_test, 10)
print('x_train.shape', x_train.shape)
print('x_test.shape', x_test.shape)
print('y_train.shape', y_train.shape)
print('y_test.shape', y_test.shape)
# ('x_train.shape', (60000, 28, 28, 1))
# ('x_test.shape', (10000, 28, 28, 1))
# ('y_train.shape', (60000, 10))
# ('y_test.shape', (10000, 10))
# Model1
model = Sequential()
model.add(Conv2D(32, kernel_size=(3, 3),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(128, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model1.png', show_shapes=True)
# Model2
model = Sequential()
model.add(Conv2D(64, kernel_size=(5, 5),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(128, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model2.png', show_shapes=True)
model.compile(loss=keras.losses.categorical_crossentropy,
optimizer=keras.optimizers.Adadelta(),
metrics=['accuracy'])
hist = model.fit(x_train, y_train,
batch_size=batch_size,
epochs=20,
validation_split=0.1,
verbose=1)
scores = model.evaluate(x_test, y_test)
print('loss={}, accuracy={}'.format(*scores))
# loss=0.0268098793532, accuracy=0.9914
plt.clf()
plt.xlabel('epochs')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['loss'], label='loss')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['val_loss'], label='val_loss')
plt.legend()
plt.savefig('model2-epochs-loss.png')
# Model3
model = Sequential()
model.add(Conv2D(32, kernel_size=(3, 3),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(128, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model3.png', show_shapes=True)
model.compile(loss=keras.losses.categorical_crossentropy,
optimizer=keras.optimizers.Adadelta(),
metrics=['accuracy'])
hist = model.fit(x_train, y_train,
batch_size=batch_size,
epochs=20,
validation_split=0.1,
verbose=1)
scores = model.evaluate(x_test, y_test)
print('loss={}, accuracy={}'.format(*scores))
# loss=0.0247306166548, accuracy=0.9926
plt.clf()
plt.xlabel('epochs')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['loss'], label='loss')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['val_loss'], label='val_loss')
plt.legend()
plt.savefig('model3-epochs-loss.png')
# Model4
model = Sequential()
model.add(Conv2D(32, kernel_size=(3, 3),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(128, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model4.png', show_shapes=True)
model.compile(loss=keras.losses.categorical_crossentropy,
optimizer=keras.optimizers.Adadelta(),
metrics=['accuracy'])
hist = model.fit(x_train, y_train,
batch_size=batch_size,
epochs=20,
validation_split=0.1,
verbose=1)
scores = model.evaluate(x_test, y_test)
print('loss={}, accuracy={}'.format(*scores))
# loss=0.0369513304536, accuracy=0.9896
plt.clf()
plt.xlabel('epochs')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['loss'], label='loss')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['val_loss'], label='val_loss')
plt.legend()
plt.savefig('model4-epochs-loss.png')
# Model5
model = Sequential()
model.add(Conv2D(32, kernel_size=(3, 3),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(MaxPooling2D(pool_size=(4, 4)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(128, activation='relu'))
model.add(Dropout(0.5))
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model5.png', show_shapes=True)
model.compile(loss=keras.losses.categorical_crossentropy,
optimizer=keras.optimizers.Adadelta(),
metrics=['accuracy'])
hist = model.fit(x_train, y_train,
batch_size=batch_size,
epochs=20,
validation_split=0.1,
verbose=1)
scores = model.evaluate(x_test, y_test)
print('loss={}, accuracy={}'.format(*scores))
# loss=0.0224432536123, accuracy=0.9927
plt.clf()
plt.xlabel('epochs')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['loss'], label='loss')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['val_loss'], label='val_loss')
plt.legend()
plt.savefig('model5-epochs-loss.png')
# Model6
model = Sequential()
model.add(Conv2D(32, kernel_size=(3, 3),
strides=(1, 1), padding='valid',
activation='relu',
input_shape=(img_rows, img_cols, 1)))
model.add(Conv2D(64, (3, 3), activation='relu'))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(num_classes, activation='softmax'))
plot_model(model, to_file='model6.png', show_shapes=True)
model.compile(loss=keras.losses.categorical_crossentropy,
optimizer=keras.optimizers.Adadelta(),
metrics=['accuracy'])
hist = model.fit(x_train, y_train,
batch_size=batch_size,
epochs=20,
validation_split=0.1,
verbose=1)
scores = model.evaluate(x_test, y_test)
print('loss={}, accuracy={}'.format(*scores))
# loss=0.0355213801761, accuracy=0.9897
plt.clf()
plt.xlabel('epochs')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['loss'], label='loss')
plt.plot(np.arange(0, len(hist.history['loss'])),
hist.history['val_loss'], label='val_loss')
plt.legend()
plt.savefig('model6-epochs-loss.png')